過去 10 年間で次世代シーケンサーの技術は、サンプル必要量を減少させたり、シーケンス読み取りの精度と深度を向上させたりするために急速に発展しました[1]。その間FANTOMコンソーシアムでは、転写開始部位 (TSS) をゲノム規模で同定する手法であるCap Analysis of Gene Expression (CAGE) テクノロジー[2] [3] の最適化および簡素化を行ってきました。技術的なバイアスを軽減し、様々なシーケンシング プラットフォーム向けに高品質でコストパフォーマンスの高いデータセットを取得してきました。
FANTOM5 コンソーシアムでは、CAP付き RNA から逆転写された cDNA をPCR増幅なしで測定できる HeliScopeCAGE[4]を開発し、哺乳動物からの多数の細胞にわたる CAGE データセットを作成しました。それらのPCRバイアスの無いCAGEデータから再現性の高いTSS解析を行い、遺伝子発現プロモーターの包括的な概要を提供しました[5]。
さらに我々は、HeliScopeCAGEをIllumina HiSeq2000/2500 シーケンサーで利用できるように改良したnon-Amplified non-Tagging Illumina Cap Analysis of Gene Expression (nAnT-iCAGE)[6] を開発しました。
FANTOM6 ではさらにそこから一本鎖cDNAをシーケンスできる一本鎖 (ss)CAGEライブラリ、少量のRNAから一本鎖cDNA をシーケンスできるLow Quantity(LQ) ssCAGE[7] の開発を導きました。ここでは、ssCAGE プロトコルのワークフローを示します(図)。
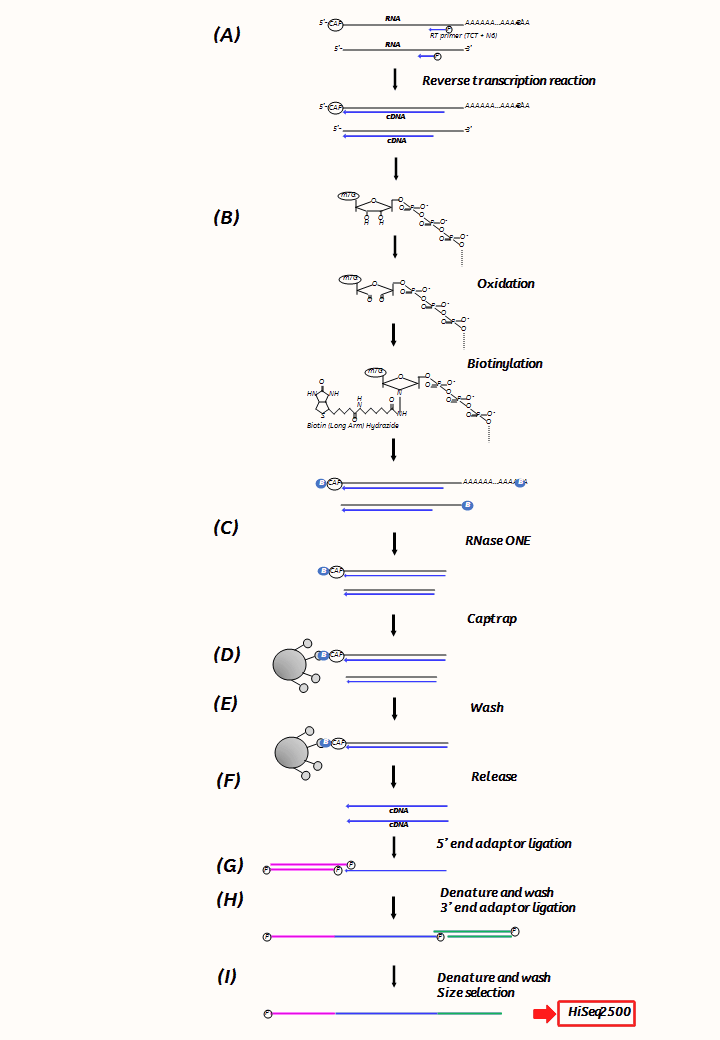
図 ssCAGEプロトコルのワークフロー
(A)逆転写反応 cDNAを SuperScript IIIおよび ランダムN6プライマーを用いて合成する。
(B)Oxidation/Biotinylation キャップ部位を過ヨウ素酸ナトリウムで酸化し、biotin (long arm) hydrazineでビオチン化する。
(C)RNaseI 消化 一本鎖 RNAを RNase Iで消化する。
(D)磁性ストレプトアビジンビーズで捕獲 ビオチン化した RNA/cDNA分子を磁性ストレプトアビジンビーズで捕獲する。
(E)非結合分子を洗い落とす。
(F)ビーズから一本鎖cDNAを外す 捕獲した RNA/cDNAから 一本鎖cDNAを熱処理により剥がし、RNase Iおよび RNase Hで消化する。
(G)5’末アダプターライゲーション ユニバーサル 5’末アダプターを 一本鎖cDNAにライゲーションする。
(H)3’末アダプターライゲーション 非結合 のdsDNA 5’末アダプターを洗い流す。インデックス配列を含む 3'末アダプターを一本鎖cDNAにライゲーションする。
(I)サイズセレクション 非結合の dsDNA 3'末アダプターを洗い流す。アダプターダイマーを完全に除去するために、SPRIビーズを用い、サイズセレクション精製を行う。 その後HiSeq2500シーケンサーで配列を読む。
Note: 本プロトコルは HiSeq2500シーケンサーでよく機能する。 NextSeq2000でも使用可能だが、パターン化フローセル上でのマルチプレックスライブラリーへのシングルインデックスは、インデックスホッピングのリスクを増大させるので推奨しない。
参考文献
- ^ H. Satam, K. Joshi, U. Mangrolia, S. Waghoo, G. Zaidi, S. Rawool, R. P. Thakare, S. Banday, A. K. Mishra, G. Das, and S. K. Malonia, Next-Generation Sequencing Technology: Current Trends and Advancements, Biol. 2023, 12(7), 997 (2023), doi: 10.3390/biology12070997
- ^ R. Kodzius, M. Kojima, H. Nishiyori, M. Nakamura, S. Fukuda, M. Tagami, D. Sasaki, K. Imamura, C. Kai, M. Harbers, Y. Hayashizaki, and P. Carninci, CAGE: cap analysis of gene expression, Nat. Methods 3, 211–222 (2006), doi: 10.1038/nmeth0306-211
- ^ H. Takahashi, T. Lassmann, M. Murata, and P. Carninci, 5' end–centered expression profiling using cap-analysis gene expression and next-generation sequencing, Nat. Protoc. 7, 542–561 (2012), doi: 10.1038/nprot.2012.005
- ^ M. Kanamori-Katayama, M. Itoh, H. Kawaji, T. Lassmann, S. Katayama, M. Kojima, N. Bertin, A. Kaiho, N. Ninomiya, C. O. Daub, P. Carninci, A. R. R. Forrest, and Y. Hayashizaki, Unamplified cap analysis of gene expression on a single-molecule sequencer, Genome Res. 21, 1150–1159 (2011), doi: 10.1101/gr.115469.110
- ^ The FANTOM Consortium and the RIKEN PMI and CLST (DGT), A promoter-level mammalian expression atlas, Nat. 507, 462–470 (2014), doi: 10.1038/nature13182
- ^ M. Murata, H. Nishiyori-Sueki, M. Kojima-Ishiyama, P. Carninci, Y. Hayashizaki, and M. Itoh, Detecting expressed genes using CAGE, Methods Mol. Biol. 1164, 67–85 (2014), doi: 10.1007/978-1-4939-0805-9_7
- ^ H. Takahashi, H. Nishiyori-Sueki, J. A. Ramilowski, M. Itoh, and P. Carninci, Low Quantity Single Strand CAGE (LQ-ssCAGE) Maps Regulatory Enhancers and Promoters, Methods Mol. Biol. 2351, 67–90 (2021), doi: 10.1007/978-1-0716-1597-3_4
