初期のCAGE法を用いたプロジェクトは、1ライブラリーあたり50~100000タグのシーケンシングを目的とし、主要な転写産物のプロモーターマッピングに対応してきました。しかしすぐに、遺伝子の数を上回る数の転写開始点(TSS)の存在が明らかになりました(マウスで230、000個以上)。さらに、特定のプロモーターの活性を定量的に解析し、統計的に評価するためには、個々のRNA分子の末端を複数回シーケンシングする必要があります。そこで我々は、当時日本で利用可能となった第2世代シーケンサー、454LifeSceinceシーケンサー[1]とCAGE法を組み合わせた「deepCAGE法」[2] [3]を開発しました。 deepCAGEは、プラスミド内でコンカテマーのクローニングをする必要がなく、全RNAサンプルからポリA(+)RNAとポリA(-)RNAの両方をとらえるために、ランダムプライマーを用いて1本目のcDNAを合成します。一般的にオリゴ-dTプライマーは、特定の解析を除き、発現解析にはお勧めできません。5’UTRのGCリッチ領域のcDNA合成を進めるためには、トレハロースとソルビトール8を加え、高温状態で逆転写反応を起こします。キャップサイトまで伸長されたcDNAはキャップトラッピング法により抽出され、鋳型となったRNAサンプルの5’末端にあたる領域が始まる部位に、クラスⅡs制限酵素Mmel(またはEcoP15I)が認識する配列を含むリンカーが結合されます。このリンカーが相補となるcDNA鎖の合成を誘導し、2本鎖cDNAが合成された後、Mmel(EcoP15I)により20-21(または27)bpのオリゴヌクレオチドが、CAGEタグ配列として切り出されます。その後Mmel(EcoP15I)の切断によってむき出しになったCAGEタグ配列の3’末端に2つめのリンカーを結合し、PCRで増幅します。454LifeSceinceシーケンサーによる解析では、タグを連結し、1回の解析で75万~100万個のCAGEタグ配列を解読しました。この大量の454-deepCAGEデータは、FANTOM4とゲノムネットワークプロジェクトによって産出されました。
Solexa 、 SOLiDによるdeepCAGE
その後発売されたSolexa(イルミナ)シーケンサーやSOLiD(ABI)シーケンサーと組み合わせることにより、deepCAGE法はさらなるコストダウンに成功しました。この技術開発では、プライマーをイルミナGA/GAII、SOLiDシーケンサー用にデザインし、厄介なコンカテマー形成のステップが排除されました。これらの次世代シーケンサーを採用したdeepCAGEの利点は以下の通りです。(a)コンカテマー形成を排除することによってPCR増幅サイクルの回数を軽減(25以上から13-15サイクルへ軽減)することが可能。これにより、PCRのバイアスを減らすことができる。(b)1回の解析でGAIIは5000万個、SOLiDは1億個以上のタグを解析できるため、1タグあたりの解析コストが削減できる。(c)CAGEタグの長さを20ntから27ntに伸ばしたことにより、マッピング率が65-70%から80-85%に改善される。また、類似の遺伝子ファミリーやゲノム領域から転写されるRNAを同定できる率が向上する。 プロモーター活性のプロファイリングには、短いタグの方が、長いものよりも理想的です。CAGEタグのような27nt程度の短いタグは、PCR反応や熱変性反応においてサイズによるバイアスを受けず、プライマー伸長による影響も受けにくい性質を持っています。一方、長くて不均一なサイズの完全長cDNAの増幅では、サイズのバイアスが起こります。たとえば、完全長cDNA の5’末端(または、ランダムプライムによる500-1000bpのcDNA5’末端)からの増幅では、そのサイズやGC含有率によって、鋳型となった転写産物の定量性にバイアスが見られます。さらに、Solexaによるシーケンシングでは、DNA分子がシーケンシングスライドに結合され、ブリッジPCRがおこなわれますが、分子がより短いほど効率的に反応が進みます(約1Kb 以下)。しかし、ほとんどの完全長cDNAのサイズはこれより大きくなります。SOLiDならびに454のアナログ増幅には やはり短いプラグメント(1kb以下)が必要なエマルジョン PCRが用いられています。このように、短いタグを使うdeepCAGE法は様々な局面で解析に有利です。また、両末端からのシーケンシングは、転写産物の末端のマッピングに重要ですが、バイアスが生じやすく、特定のプロモーターにおける転写活性の解析には適しません。
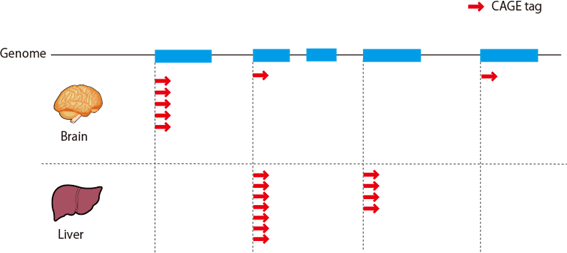
図1: deepCAGEによって同定された転写開始点とその転写産物の量
参考文献
- ^ Maeda, N. et al. Development of a DNA barcode tagging method for monitoring dynamic changes in gene expression by using an ultra high-throughput sequencer. Biotechniques 45, 95-7 (2008), doi: 10.2144/000112814
- ^ de Hoon M, Hayashizaki Y Deep cap analysis gene expression (CAGE): genome-wide identification of promoters, quantification of their expression, and network inference. Biotechniques 2008 Apr;44(5):627-8, 630, 632, doi: 10.2144/000112802
- ^ Valen et al. Genome-wide detection and analysis of hippocampus core promoters using DeepCAGE Genome Research 19:255-265 2009, doi: 10.1101/gr.084541.108
